2.2 Group by individual
個体間のassociationを表したデータとしては、以下のようにある時点・場所において確認された個体を各行に0/1で記録した”group by individual”と呼ばれる形式のものがよく用いられる。
例えば、以下は金華山島\(B_1\)群で2021年交尾期の各観察日(date)に9歳以上のメスが観察されたか否かを示したものである。
presence <- read_csv("data/presence_demo.csv")
presence %>%
datatable(rownames = FALSE,
options = list(scrollX = 50),
filter = "top")このようなデータでは、同じグループで観察された個体をassociateしていたとみなすことが多い(=gambit of the group)(Farine & Whitehead, 2015)。Group by individualの形式のデータでは、以下のような指標で個体間の強さを表すことが多い。なお、各指標の詳細については W. J. E. Hoppitt & Farine (2018) を参照。
- \(x\): 個体\(a\)と\(b\)が一緒に観察された回数
- \(y_a\): \(a\)だけが確認された回数
- \(y_b\): \(b\)だけが確認された回数
- \(y_{ab}\): \(a\)と\(b\)が共に観察されたが、associateはしてない回数(今回の例では存在しない)
SRI: simple ratio index
\(= x/(y_a + y_b + y_{ab} + x)\)
▶ 単純に一緒に観察された割合。観察ミス(個体がいたにもかかわらずいなかったと記録してしまうこと)が少ない場合にはこちらで問題ない。
HWI: half-weight index
\(= x/(\frac{1}{2}(y_a + y_b) + y_{ab} + x)\)
▶ 観察ミスが多いと考えられる場合、補正を行う。
HWIG (Godde et al., 2013)
\(= HWI_{ab} \times \frac{\sum_i \sum_j HWI_{ij}}{\sum_i HWI_{ai} \times \sum_i HWI_{bi}}\)
▶ それぞれの個体のgregariousnessを考慮したHWI。互いのgregariousnessを考慮したとき、ランダムにassociateしているのであれば1になり、1よりおおきければランダムの場合よりもよくassociateしていることになる。
asnipeパッケージのget_network()関数では、“group by individual”形式のデータからSRIとHWIを算出し、マトリックスを作成することが可能である。
presence_mat <- get_network(presence %>%
## 日付の列は除く
dplyr::select(-date),
## gambit of the group
data_format = "GBI",
## "HWI"の場合は、"HWI"
association_index = "SRI") ## Generating 15 x 15 matrix| Mik | Kil | Koh | Aka | Ntr | Ten | Tot | Hen | Hot | Mei | Ako | Kor | Mal | Kit | Kun | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Mik | 0.00 | 0.43 | 0.45 | 0.63 | 0.41 | 0.46 | 0.41 | 0.39 | 0.32 | 0.43 | 0.45 | 0.43 | 0.32 | 0.42 | 0.42 |
| Kil | 0.43 | 0.00 | 0.98 | 0.45 | 0.93 | 0.95 | 0.95 | 0.94 | 0.80 | 0.98 | 0.80 | 0.98 | 0.88 | 0.95 | 0.95 |
| Koh | 0.45 | 0.98 | 0.00 | 0.45 | 0.95 | 0.95 | 0.95 | 0.94 | 0.78 | 0.98 | 0.80 | 0.98 | 0.86 | 0.95 | 0.95 |
| Aka | 0.63 | 0.45 | 0.45 | 0.00 | 0.44 | 0.46 | 0.42 | 0.42 | 0.39 | 0.43 | 0.53 | 0.43 | 0.35 | 0.43 | 0.41 |
| Ntr | 0.41 | 0.93 | 0.95 | 0.44 | 0.00 | 0.90 | 0.90 | 0.89 | 0.76 | 0.93 | 0.77 | 0.93 | 0.86 | 0.93 | 0.93 |
| Ten | 0.46 | 0.95 | 0.95 | 0.46 | 0.90 | 0.00 | 0.93 | 0.89 | 0.76 | 0.95 | 0.77 | 0.95 | 0.83 | 0.93 | 0.93 |
| Tot | 0.41 | 0.95 | 0.95 | 0.42 | 0.90 | 0.93 | 0.00 | 0.89 | 0.78 | 0.95 | 0.77 | 0.95 | 0.86 | 0.95 | 0.95 |
| Hen | 0.39 | 0.94 | 0.94 | 0.42 | 0.89 | 0.89 | 0.89 | 0.00 | 0.81 | 0.92 | 0.76 | 0.92 | 0.91 | 0.92 | 0.92 |
| Hot | 0.32 | 0.80 | 0.78 | 0.39 | 0.76 | 0.76 | 0.78 | 0.81 | 0.00 | 0.78 | 0.65 | 0.78 | 0.79 | 0.78 | 0.78 |
| Mei | 0.43 | 0.98 | 0.98 | 0.43 | 0.93 | 0.95 | 0.95 | 0.92 | 0.78 | 0.00 | 0.78 | 0.98 | 0.86 | 0.95 | 0.95 |
| Ako | 0.45 | 0.80 | 0.80 | 0.53 | 0.77 | 0.77 | 0.77 | 0.76 | 0.65 | 0.78 | 0.00 | 0.78 | 0.70 | 0.78 | 0.78 |
| Kor | 0.43 | 0.98 | 0.98 | 0.43 | 0.93 | 0.95 | 0.95 | 0.92 | 0.78 | 0.98 | 0.78 | 0.00 | 0.88 | 0.95 | 0.95 |
| Mal | 0.32 | 0.88 | 0.86 | 0.35 | 0.86 | 0.83 | 0.86 | 0.91 | 0.79 | 0.86 | 0.70 | 0.88 | 0.00 | 0.88 | 0.88 |
| Kit | 0.42 | 0.95 | 0.95 | 0.43 | 0.93 | 0.93 | 0.95 | 0.92 | 0.78 | 0.95 | 0.78 | 0.95 | 0.88 | 0.00 | 0.98 |
| Kun | 0.42 | 0.95 | 0.95 | 0.41 | 0.93 | 0.93 | 0.95 | 0.92 | 0.78 | 0.95 | 0.78 | 0.95 | 0.88 | 0.98 | 0.00 |
HWIGはhwigパッケージのcalc_hwig()関数で算出することができる。
## まずHWIを算出
presence_mat_HWI <- get_network(presence %>%
dplyr::select(-date),
data_format = "GBI",
association_index = "HWI")## Generating 15 x 15 matrix## HWIGの算出
presence_mat_HWIG <- presence_mat_HWI %>%
## data.table形式に直す必要
as.data.table() %>%
calc_hwig() %>%
as.matrix()
rownames(presence_mat_HWIG) <- rownames(presence_mat_HWI)| Mik | Kil | Koh | Aka | Ntr | Ten | Tot | Hen | Hot | Mei | Ako | Kor | Mal | Kit | Kun | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Mik | 0.00 | 0.51 | 0.52 | 0.96 | 0.50 | 0.54 | 0.49 | 0.48 | 0.45 | 0.51 | 0.57 | 0.51 | 0.43 | 0.50 | 0.50 |
| Kil | 0.51 | 0.00 | 0.54 | 0.50 | 0.54 | 0.54 | 0.54 | 0.54 | 0.54 | 0.55 | 0.53 | 0.55 | 0.55 | 0.54 | 0.54 |
| Koh | 0.52 | 0.54 | 0.00 | 0.50 | 0.55 | 0.54 | 0.54 | 0.54 | 0.53 | 0.55 | 0.53 | 0.55 | 0.54 | 0.54 | 0.54 |
| Aka | 0.96 | 0.50 | 0.50 | 0.00 | 0.51 | 0.52 | 0.49 | 0.49 | 0.50 | 0.49 | 0.62 | 0.49 | 0.45 | 0.49 | 0.48 |
| Ntr | 0.50 | 0.54 | 0.55 | 0.51 | 0.00 | 0.54 | 0.54 | 0.54 | 0.54 | 0.54 | 0.53 | 0.54 | 0.55 | 0.54 | 0.55 |
| Ten | 0.54 | 0.54 | 0.54 | 0.52 | 0.54 | 0.00 | 0.54 | 0.54 | 0.53 | 0.55 | 0.53 | 0.54 | 0.54 | 0.54 | 0.54 |
| Tot | 0.49 | 0.54 | 0.54 | 0.49 | 0.54 | 0.54 | 0.00 | 0.54 | 0.54 | 0.55 | 0.53 | 0.55 | 0.54 | 0.55 | 0.55 |
| Hen | 0.48 | 0.54 | 0.54 | 0.49 | 0.54 | 0.54 | 0.54 | 0.00 | 0.56 | 0.54 | 0.53 | 0.54 | 0.57 | 0.54 | 0.54 |
| Hot | 0.45 | 0.54 | 0.53 | 0.50 | 0.54 | 0.53 | 0.54 | 0.56 | 0.00 | 0.54 | 0.52 | 0.54 | 0.57 | 0.54 | 0.54 |
| Mei | 0.51 | 0.55 | 0.55 | 0.49 | 0.54 | 0.55 | 0.55 | 0.54 | 0.54 | 0.00 | 0.53 | 0.55 | 0.54 | 0.54 | 0.54 |
| Ako | 0.57 | 0.53 | 0.53 | 0.62 | 0.53 | 0.53 | 0.53 | 0.53 | 0.52 | 0.53 | 0.00 | 0.53 | 0.52 | 0.53 | 0.53 |
| Kor | 0.51 | 0.55 | 0.55 | 0.49 | 0.54 | 0.54 | 0.55 | 0.54 | 0.54 | 0.55 | 0.53 | 0.00 | 0.55 | 0.54 | 0.54 |
| Mal | 0.43 | 0.55 | 0.54 | 0.45 | 0.55 | 0.54 | 0.54 | 0.57 | 0.57 | 0.54 | 0.52 | 0.55 | 0.00 | 0.55 | 0.55 |
| Kit | 0.50 | 0.54 | 0.54 | 0.49 | 0.54 | 0.54 | 0.55 | 0.54 | 0.54 | 0.54 | 0.53 | 0.54 | 0.55 | 0.00 | 0.55 |
| Kun | 0.50 | 0.54 | 0.54 | 0.48 | 0.55 | 0.54 | 0.55 | 0.54 | 0.54 | 0.54 | 0.53 | 0.54 | 0.55 | 0.55 | 0.00 |
得られたSRIの隣接行列についてグラフを描画すると図2.2のようになる。ノードの大きさは年齢、色は順位を、辺の太さはHWIGを表す。MikとAkaは他の個体と一緒にいることが少ないことが分かるだろう(年齢も影響しそう?)。順位の高い個体はネットワークの中心にいることが多そう?
age <- c(18,17,15,15,13,13,13,13,13,13,12,11,11,10,9)
rank2 <- c(16,1,3,9,7,8,12,13,14,17,10,6,15,2,5)
set.seed(129)
presence_mat %>%
as_tbl_graph(directed = FALSE) %>%
ggraph(layout = "nicely")+
# 曲線のエッジ、weightを太さに
geom_edge_link(aes(width = weight),
## 透明度(alpha)と色(color)指定
alpha =0.7, color = "grey67")+
# エッジの太さの範囲を決める
scale_edge_width(range = c(0,1.5))+
## 四角のノード。大きさは年齢によって変化するとする。
geom_node_point(aes(size = age, color = rank2),shape = 16)+
## ノードのラベルは個体名
scale_size(range = c(2,5))+
geom_node_text(aes(label = name),
## ノードと重ならないようにする
repel=TRUE, size =6)+
theme_graph()+
## 縦横比
theme(aspect.ratio = 0.7)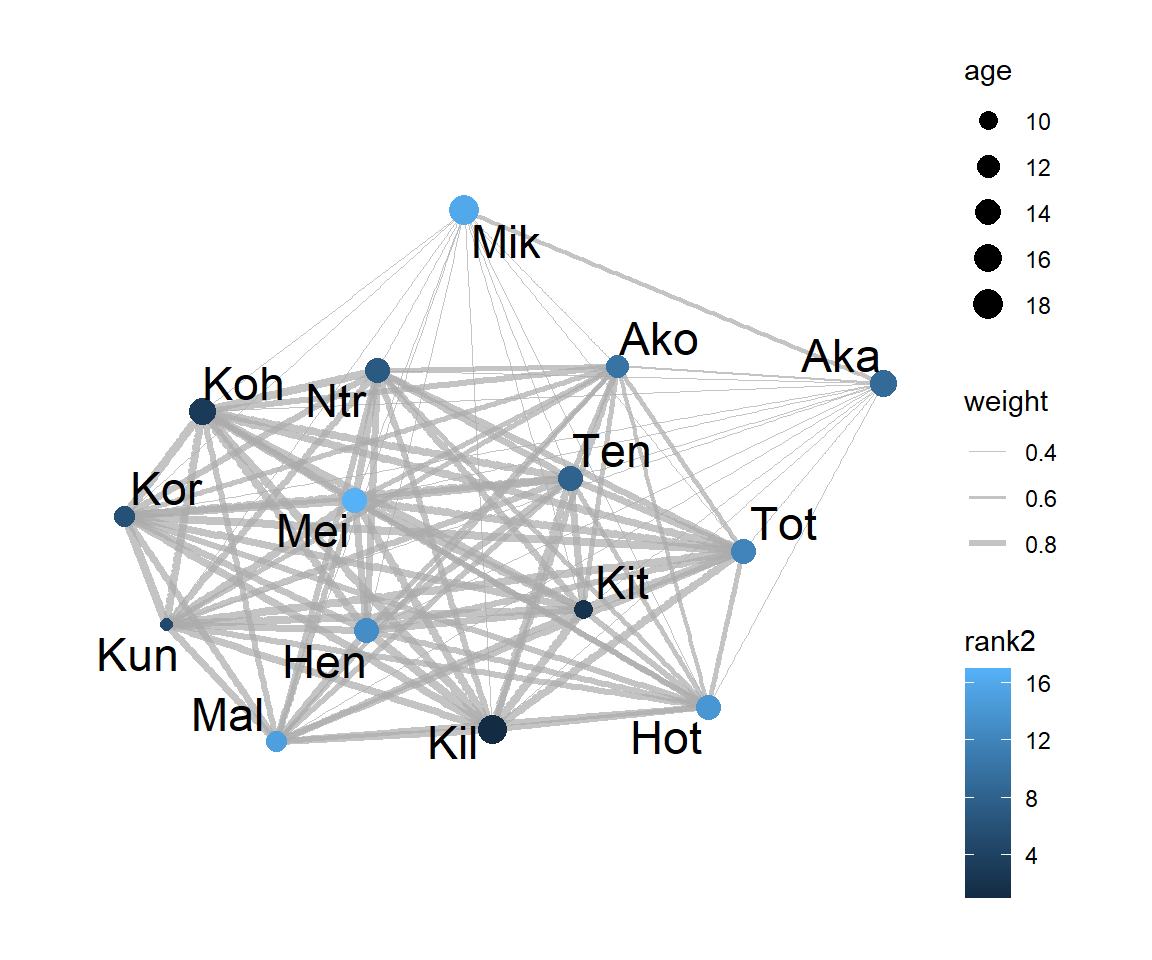
図2.2: SRIネットワークのグラフ