2 データの読み込みとマトリックスの作成
R上でSNAを行うためには、個体間の関係データを隣接行列にしなければならないことが多い。しかし多くの場合、個体間のassociationの生データは隣接行列の形では表されていない。例えば個体追跡のデータでは、以下(表2.1)のように1分ごとに毛づくろい相手のIDを記録したものをまずExcelファイルなどに記入する、というようなことが多いだろう。ここでは、R上でこのような生データから隣接行列を作成する方法を学ぶ。
| min | groomer | groomee |
|---|---|---|
| 1 | A | B |
| 2 | A | B |
| 3 | A | C |
| 4 | B | A |
| 5 | B | A |
2.1 2個体間の交渉を記録したデータフレーム
ここでは、2個体間の交渉(例えば、毛づくろいなど)を記録したデータフレームから隣接行列を作る。
例えば、以下は金華山島\(B_1\)群で2018年時点で6歳以上のメスを個体追跡した際のデータであり、1分間の瞬間サンプリングで以下を記録したデータフレームである。
no_focal: 個体追跡セッション番号
subject: 追跡個体名
time: 個体追跡開始からの経過時間
activity: 個体の活動(G: 毛づくろい、R: 休息、F: 採食、M: 移動、O: その他)
TG: 樹上(T)にいたか地上(G)にいたか
groomer:activityが毛づくろいだったときのgroomer
geoomee:activityが毛づくろいだったときのgroomer
groom <- read_csv("data/focal_demo.csv")
groom %>%
head(50) %>%
datatable(rownames = FALSE,
options = list(scrollX = 50),
filter = "top")追跡したメスのIDは以下のとおりである。
adult <- c("Kil","Kit","Koh","Kur","Kun","Kor","Ntr",
"Ten","Aka","Ako","Tam","Tot","Hen","Hot",
"Mal","Mik","Mei")このデータをもとに、個体追跡中にメス間が毛づくろいをした頻度を示したマトリックスを作成する。
ここで、頻度は以下の式で表すものとする。
- \(G_{AB}\): \(A\)から\(B\)への毛づくろい頻度
- \(x_{AB}\): \(A\)から\(B\)への毛づくろいが確認された瞬間サンプリングポイント数
- \(y_A\): \(A\)の個体追跡時間(瞬間サンプリングポイント総数)
- \(y_B\): \(B\)の個体追跡時間(瞬間サンプリングポイント総数)
\[
G_{AB} = \frac{x_{AB}}{y_{A} + y_{B}}
\]
分母となる各個体の個体追跡時間(\(y_A, y_B, ...\))を算出する。
groom %>%
group_by(subject) %>%
summarise(duration = n()) -> duration
duration %>%
datatable(rownames = FALSE,
options = list(scrollX = 50),
filter = "top")追跡個体がのactivityが毛づくろいで、かつ地上にいたポイントのみを抽出する。
また、毛づくろい相手が大人だったのみを抽出。
groom_G <- groom %>%
## 地上かつ毛づくろいのみ抽出
filter(activity == "G") %>%
filter(Groomer %in% adult & Groomee %in% adult)毛づくろい頻度はANTsパッケージのdf.to.mat()関数で以下のように求められる。
groom_mat <- df.to.mat(groom_G,
## 行動の行い手を表す列
actor = "Groomer",
## 行動の受け手を表す列
receiver = "Groomee",
## 追跡時間
tobs = duration$duration,
## 交渉の方向を考慮するか。FALSEでする。
sym = FALSE)| Aka | Ako | Hen | Hot | Kil | Kit | Koh | Kor | Kun | Kur | Mal | Mei | Mik | Ntr | Tam | Ten | Tot | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Aka | 0.00 | 0.04 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Ako | 0.07 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 |
| Hen | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Hot | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 |
| Kil | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Kit | 0.00 | 0.00 | 0.00 | 0.00 | 0.02 | 0.00 | 0.01 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Koh | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.01 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Kor | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.02 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 |
| Kun | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 |
| Kur | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.01 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Mal | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.02 | 0.00 | 0.00 | 0.00 | 0.01 |
| Mei | 0.01 | 0.01 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Mik | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.03 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Ntr | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.01 | 0.00 |
| Tam | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 |
| Ten | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 |
| Tot | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 |
もし交渉の方向性を考慮しない場合は、sym = TRUEとすればよい。
groom_mat_b <- df.to.mat(groom_G,
## 行動の行い手を表す列
actor = "Groomer",
## 行動の受け手を表す列
receiver = "Groomee",
## 追跡時間
tobs = duration$duration,
sym = TRUE)| Aka | Ako | Hen | Hot | Kil | Kit | Koh | Kor | Kun | Kur | Mal | Mei | Mik | Ntr | Tam | Ten | Tot | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Aka | 0.00 | 0.11 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Ako | 0.11 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.02 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 |
| Hen | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.01 | 0.00 | 0.00 | 0.01 | 0.01 | 0.00 |
| Hot | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 |
| Kil | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.04 | 0.02 | 0.00 | 0.01 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Kit | 0.00 | 0.00 | 0.00 | 0.00 | 0.04 | 0.00 | 0.03 | 0.01 | 0.00 | 0.02 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| Koh | 0.00 | 0.00 | 0.00 | 0.00 | 0.02 | 0.03 | 0.00 | 0.02 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 |
| Kor | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.01 | 0.02 | 0.00 | 0.01 | 0.02 | 0.00 | 0.00 | 0.01 | 0.01 | 0.00 | 0.01 | 0.01 |
| Kun | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 |
| Kur | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.02 | 0.00 | 0.02 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 |
| Mal | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.04 | 0.00 | 0.00 | 0.00 | 0.01 |
| Mei | 0.01 | 0.02 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.01 | 0.00 | 0.00 | 0.00 |
| Mik | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.01 | 0.00 | 0.04 | 0.01 | 0.00 | 0.01 | 0.01 | 0.00 | 0.00 |
| Ntr | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 | 0.01 | 0.01 | 0.00 | 0.00 | 0.01 | 0.01 |
| Tam | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.02 |
| Ten | 0.00 | 0.01 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 |
| Tot | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.01 | 0.02 | 0.00 | 0.00 |
グラフを描画すると図2.1のようになる。ノードの大きさは順位を、辺の太さは毛づくろい頻度を表す。血縁個体同士(同じアルファベットで始まる個体)は交渉頻度が高く、近くに配置されていることが分かる。順位の低い個体がネットワークの周辺にいることが多いことも分かるだろう。
## 順位
rank <- c(9,10,13,14,1,2,3,6,5,4,15,17,16,7,11,8,12)
groom_mat_b %>%
as_tbl_graph(directed = FALSE) %>%
## 順位の情報を入れる
mutate(rank = rank) %>%
ggraph(layout = "nicely")+
# 曲線のエッジ、weightを太さに
geom_edge_fan(aes(width = weight),
## 透明度(alpha)と色(color)指定
alpha =0.7, color = "grey60")+
# エッジの太さの範囲を決める
scale_edge_width(range = c(0.3,3))+
## 四角のノード。大きさは年齢によって変化するとする。
geom_node_point(aes(size = -rank), shape = 18)+
## ノードのラベルは個体名
scale_size(range = c(1,5))+
geom_node_text(aes(label = name),
## ノードと重ならないようにする
repel=TRUE, size =6)+
theme_graph()+
## 縦横比
theme(aspect.ratio = 0.7)+
labs(width = "毛づくろい頻度")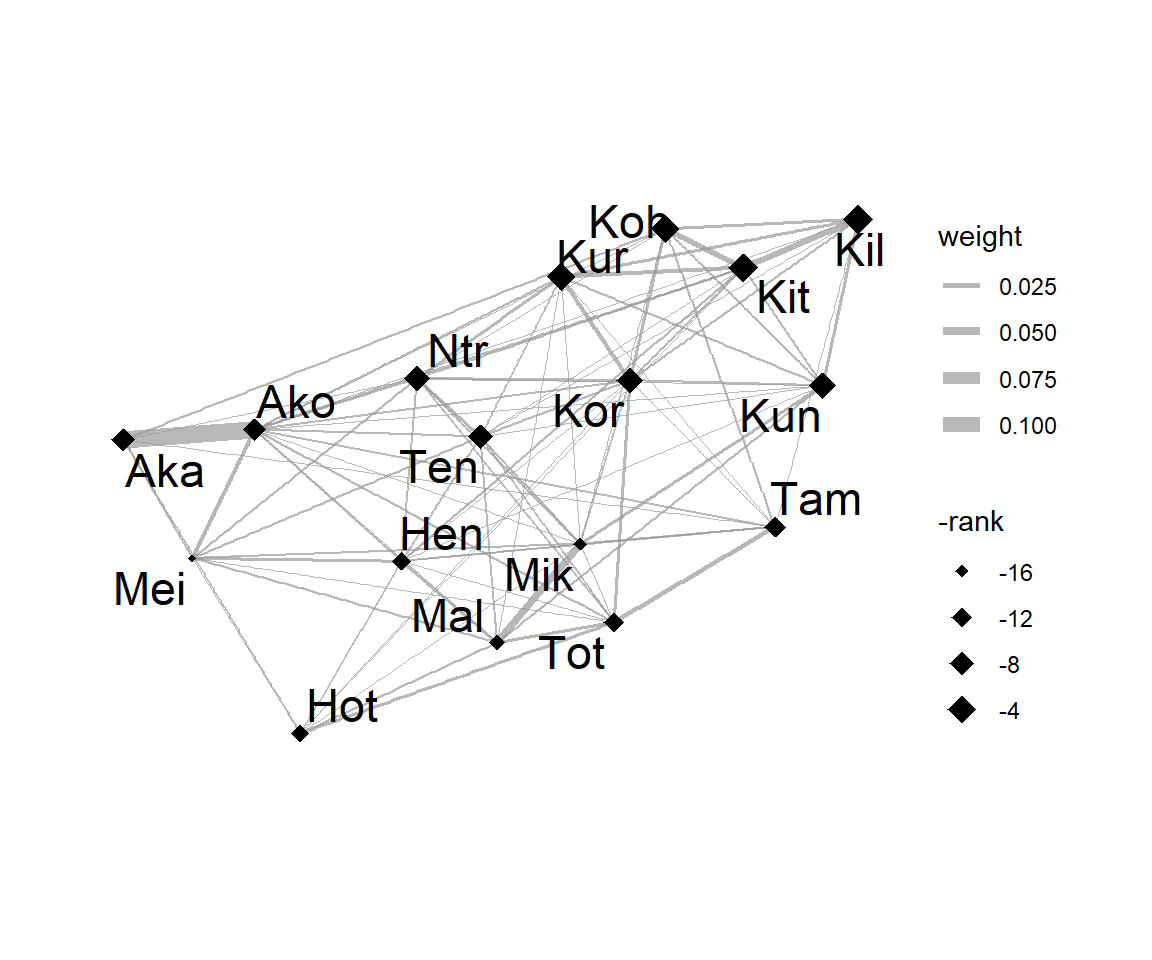
図2.1: 毛づくろい頻度ネットワークのグラフ